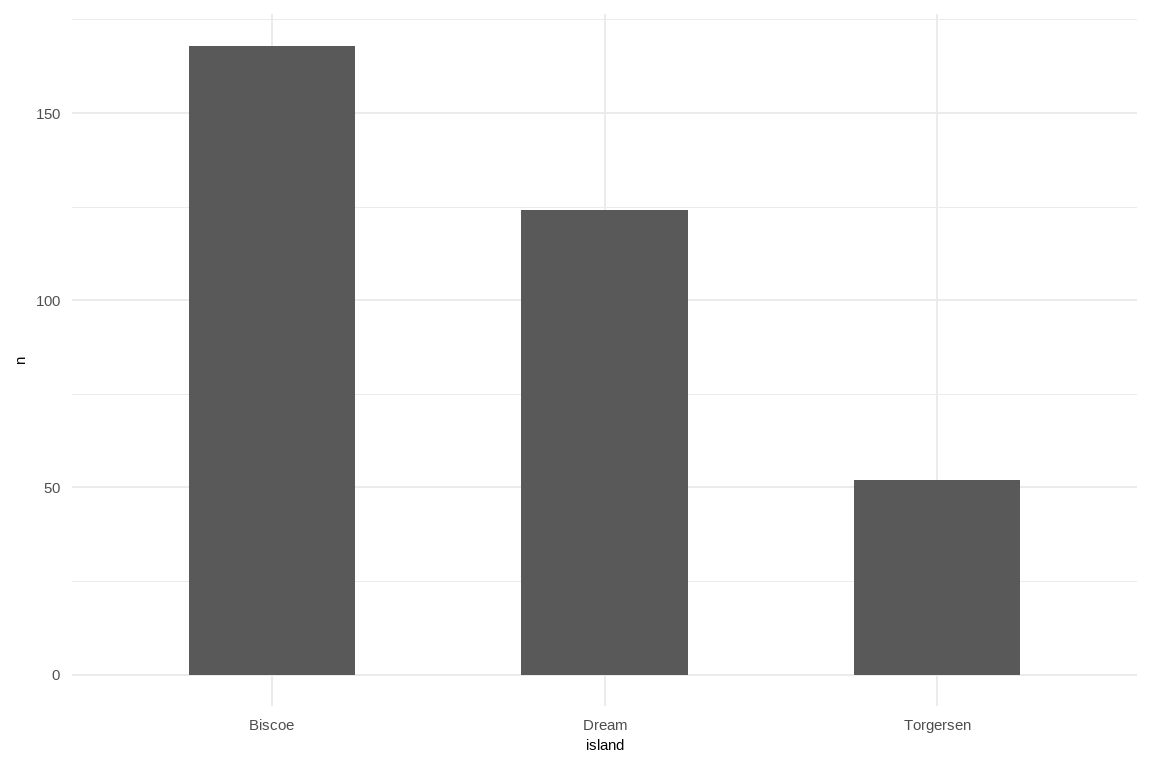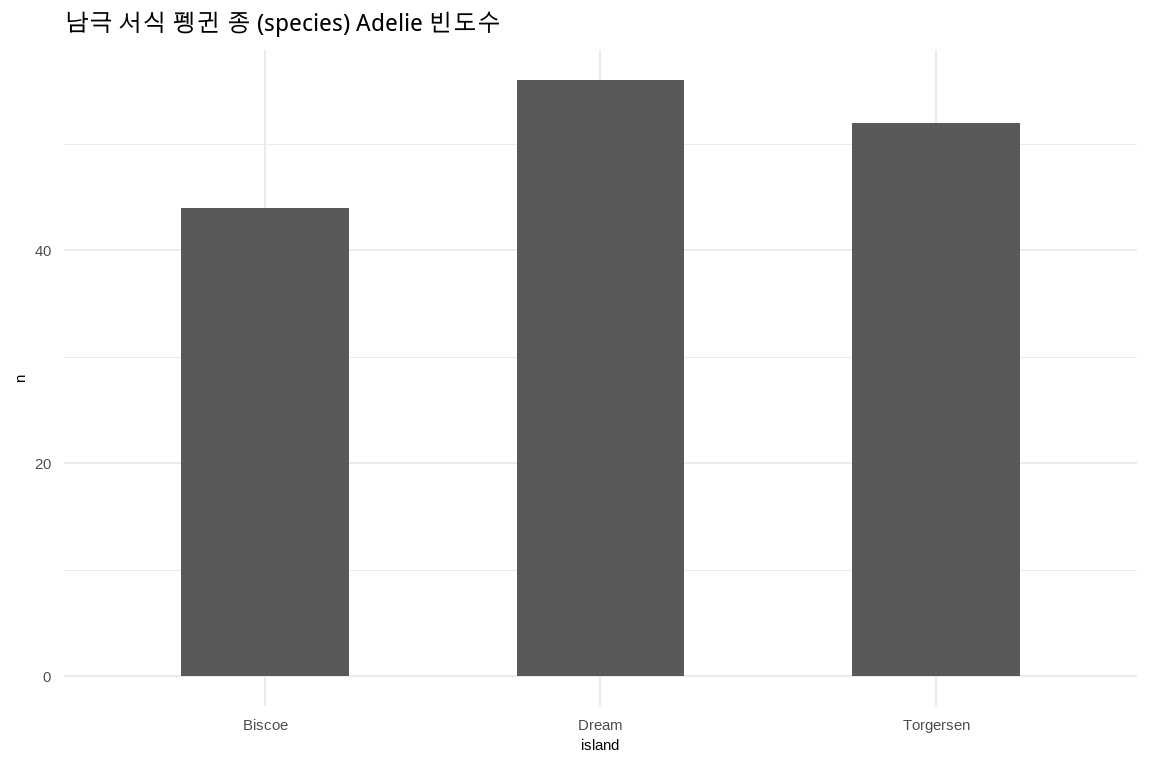library(dplyr)
library(palmerpenguins)
filter_na <- function(dataframe, col_name) {
col_quo = enquo(col_name) # 신관제거(defuse)
dataframe %>%
select(species, island, sex, year, body_mass_g) |>
filter(is.na(!!col_quo)) # 주입(inject)
}
# 사용방법
penguins %>% filter_na(sex)
#> # A tibble: 11 × 5
#> species island sex year body_mass_g
#> <fct> <fct> <fct> <int> <int>
#> 1 Adelie Torgersen <NA> 2007 NA
#> 2 Adelie Torgersen <NA> 2007 3475
#> 3 Adelie Torgersen <NA> 2007 4250
#> 4 Adelie Torgersen <NA> 2007 3300
#> 5 Adelie Torgersen <NA> 2007 3700
#> 6 Adelie Dream <NA> 2007 2975
#> # ℹ 5 more rows17 데이터 과학 함수
일반 프로그래밍 언어 함수와 tidyverse 데이터 과학 함수에서 차이점이 발견되는데 그 이유는 개발된 목적과 사용 환경 차이에 기인한다. C/C++, 파이썬, 자바스크립트와 같은 프로그래밍 언어 함수는 광범위한 프로그래밍 작업에 적합하도록 설계된 반면, tidyverse 함수는 R 언어에서 데이터 조작과 시각화, 모형 개발에 특화되었다.
따라서, R 언어 tidyverse 프레임워크는 일반 프로그래밍 언어가 갖는 함수의 장점은 차용하고 불편한 점은 개선하여 데이터 과학 업무에 적합한 함수 개발 및 활용 체계를 구축하게 되었다. 특히 tidyverse를 구성하는 dplyr, ggplot2 같은 패키지는 깔끔한 데이터(‘tidy’) 형식을 공통 기반으로 삼고 있어 데이터 과학 작업을 더 직관적이고 효율적으로 수행할 수 있다.
단순한 데이터 과학 업무는 인터랙티브하게 콘솔에서 작업을 수행하지만, 난이도가 올라가고 좀 더 복잡한 작업은 스크립트를 작성한다. 스크립트 작업을 여러 번 반복하게 되면 복사하여 붙여넣기를 자주하게 된다. 그런 과정에서 뜻하지 않은 실수를 누구나 겪게 된다. 이제 함수를 작성하여 이러한 실수를 줄이고 생산성을 높여나갈 시점이다. 함수를 작성하면 복사하여 붙여넣기를 사용할 때보다 몇 가지 큰 이점이 있다.
- 기억하기 좋고 연상적인 함수명은 코드 가독성을 높인다.
- 복사하여 붙여넣기에 따른 버그를 없앨 수 있다.
- 각종 요구사항의 변경이 있더라도 한두 곳만 코드를 수정하면 문제를 해결할 수 있어 생산성이 올라간다.
- 함수로 기능을 구현해 놓으면 나중에 패키지를 제작할 때 혹은 다른 프로젝트에 재사용이 용이하여 작성한 지적 산출물 공유를 촉진시킨다.
즉, 함수를 작성하는 이유는 반복되는 중복 문제를 해결하고 추상화를 통해 더 복잡한 작업을 가능하게 만들기 위해서이다. 거의 모든 데이터 과학 분야에 함수가 사용되지만 다음 세 가지 사례를 중점적으로 살펴보자.
- 벡터를 입력으로 받아 벡터를 반환하는 함수.
- 데이터프레임을 입력받아 데이터프레임을 반환하는 함수.
- 데이터프레임을 입력받아 그래프를 반환하는 함수.
17.1 신관제거 주입 패턴
tidy evaluation에서 신관제거 주입(Defuse-and-Inject) 패턴을 통해 데이터프레임을 다루는 dplyr 패키지와 그래프 문법에 따른 시각화를 수행하는 ggplot2 패키지에 함수를 직관적으로 적용시킬 수 있다. 신관제거(defuse)는 기본적으로 표현식의 평가를 지연시켜 바로 실행되는 것을 막는 역할을 수행한다. 이런 기능을 통해 환경의 맥락을 유지하는 역할을 한다. 주입(injection)은 포획되거나 신관제거된 표현식을 다른 맥락에서 평가하거나 다른 표현식에 주입하는 개념이다. 신관제거에 enquo()가 사용되었다면, 주입에는 !! (뱅뱅이라고 읽음) 연산자를 사용하여 다른 함수 내부에서 평가되어 실행되는 역할을 수행한다.
filter_na() 함수는 데이터프레임과 칼럼명을 인자로 전달받아 칼럼명에 결측값이 있는 행만 추출하여 반환하는 역할을 수행한다. 이를 위해 칼럼명을 신관제거하여 col_quo 표현식으로 지연시킨 후에 !!col_quo에 주입시켜 평가 작업을 수행하여 원하는 결과를 반환한다.
17.2 역사
tidyverse는 데이터 마스킹(data-masking) 방식을 ggplot2, dplyr 패키지에 도입했지만, 결국 rlang 패키지에 자체 프로그래밍 프레임워크를 장착했다. rlang 패키지의 신관제거 주입(Defuse-and-Inject) 패턴에 이르는 과정은 이전의 다양한 시도를 통해 학습된 배움의 과정이었다.
- S언어에서
attach()함수로 데이터 범위 개념을 도입했다. (Becker 2018) - S언어로 모형 함수에 데이터 마스킹 공식을 도입했다. (Chambers 와/과 Hastie 1992)
- 피터 델가드(Peter Delgaard)가
frametools패키지를 1997년 작성했고 나중에base::transform(),base::subset()함수로 베이스 R에 채택됐다. - 루크 티어니(Luke Tierney)가 원래 환경을 추적하기 위해 공식을 2000년에 변경했고 R 1.1.0에 반영되었으며
Quosures의 모태가 되었다. - 2001년 루크 티어니는
base::with()를 소개했다. -
dplyr패키지가 2014년 첫선을 보였고, 2017년rlang패키지에tidy eval이 구현되며quosure, 암묵적 주입(implicit injection), 데이터 대명사(data pronouns) 개념이 소개됐다. - 2019년
rlang0.4.0에Defuse-and-Inject패턴을 단순화한{{}}이 도입되어 직관적으로 코드를 작성하게 되었다.
데이터 분석에서 빈도수가 높은 작업을 Base R과 dplyr 패키지를 사용한 사례를 다음과 같이 비교하면 S언어에서 현재까지 이뤄낸 발전이 가시적으로 다가온다.
| 작업 | Base R | dplyr |
|---|---|---|
| 행 필터링 (Filter) | subset(data, condition) |
data %>% filter(condition) |
| 특정 칼럼 선택 (Select) | data[, c("col1", "col2")] |
data %>% select(col1, col2) |
| 그룹별 집계작업 | aggregate(. ~ grouping_var, data, FUN = mean) |
data %>% group_by(grouping_var) %>% summarize(new_col = mean(col_name)) |
| 조인(Join) | merge(data1, data2, by = "key_column") |
data1 %>% inner_join(data2, by = "key_column") |
| 칼럼 추가 | transform(data, new_col = some_func(existing_col)) |
data %>% mutate(new_col = some_func(existing_col)) |
| 행 결합 | rbind(data1, data2) |
bind_rows(data1, data2) |
| 칼럼 결합 | cbind(data1, data2) |
bind_cols(data1, data2) |
| 정렬 | data[order(data$col_name), ] |
data %>% arrange(col_name) |
17.2.1 attach() 함수
데이터프레임에 attach() 함수를 사용하면 데이터프레임을 구성하는 칼럼이 벡터로 작업 환경에서 바로 접근하여 작업을 수행할 수 있다. penguins 데이터프레임을 attach()한 결과 bill_depth_mm 벡터가 작업 환경에서 바로 접근하여 평균값을 계산할 수 있게 되었다. 작업을 완료한 후에는 detach()를 사용해서 작업 환경에서 제거한다.
17.2.2 with() 함수
attach() 함수는 편리한 장점이 있지만, 데이터프레임 변수명과 함수명, 또 다른 작업에서 나온 객체명과 충돌이 발생할 경우 전혀 생각하지 못한 문제가 발생할 수 있다. 따라서, 격리를 통해 문제를 단순화하는 것이 필요하다. 이를 위해서 with() 함수를 사용하게 되면 데이터프레임에 속한 칼럼명을 명시하지 않더라도 간결하게 데이터 분석 작업을 이어나갈 수 있다.
library(palmerpenguins)
with(data = penguins,
expr = mean(bill_depth_mm, na.rm = TRUE))
#> [1] 17.15117
17.2.3 aggregate() 함수
Base R에서 지원되는 aggregate() 함수를 사용해서 동일한 결과를 얻을 수 있다. aggregate() 함수는 with()와 지향점은 유사하지만 구현 방식에서 다소 차이가 난다.
aggregate(bill_depth_mm ~ 1,
data = penguins,
FUN = mean,
na.rm = TRUE)
#> bill_depth_mm
#> 1 17.1511717.3 데이터 마스킹
R에서의 데이터 마스킹(Data Masking)은 tidyverse 생태계에서 데이터 문법을 담당하는 dplyr 패키지에서 핵심적인 개념이다. 데이터 마스킹을 사용하면 데이터프레임 칼럼을 $, [[ ]]를 사용하지 않고도 칼럼명으로 직접 참조할 수 있어, 데이터를 조작하고 변환할 때 훨씬 직관적이고 가독성 높은 코드를 작성할 수 있다.
penguins 데이터프레임의 species를 데이터 마스킹 없이 조작하려면 penguins$species 혹은 penguins[['species']]와 같이 구문을 작성해야 하지만 데이터 마스킹을 사용하면 species만으로 충분하다.
library(dplyr)
library(palmerpenguins)
# 데이터 마스킹을 사용하여 펭귄 종(species)이 "Adelie"인 것만 추출한다.
penguins %>% filter(species == "Adelie")
#> # A tibble: 152 × 8
#> species island bill_length_mm bill_depth_mm flipper_length_mm
#> <fct> <fct> <dbl> <dbl> <int>
#> 1 Adelie Torgersen 39.1 18.7 181
#> 2 Adelie Torgersen 39.5 17.4 186
#> 3 Adelie Torgersen 40.3 18 195
#> 4 Adelie Torgersen NA NA NA
#> 5 Adelie Torgersen 36.7 19.3 193
#> 6 Adelie Torgersen 39.3 20.6 190
#> # ℹ 146 more rows
#> # ℹ 3 more variables: body_mass_g <int>, sex <fct>, year <int>dplyr 함수의 데이터 마스킹은 비표준 평가(Non-standard evaluation, NSE)라는 개념에 기반을 두는데, 표현식을 캡처하고 난 후 바로 실행되지 않고 제공된 데이터의 맥락 내에서 평가가 이루어진다. 데이터 마스킹은 강력하며 깔끔한 구문을 제공하지만, 칼럼명과 충돌할 수 있는 환경의 변수 이름이 있을 때 예기치 않은 방식으로 동작한다. 모호한 상황이 발생할 때 항상 다음과 같은 방식으로 .data$column_name을 사용함으로써 데이터 마스킹 재정의(Overriding)를 통해 명확히 한다.
species <- "Chinstrap"
penguins %>%
filter(.data$species == "Adelie")
#> # A tibble: 152 × 8
#> species island bill_length_mm bill_depth_mm flipper_length_mm
#> <fct> <fct> <dbl> <dbl> <int>
#> 1 Adelie Torgersen 39.1 18.7 181
#> 2 Adelie Torgersen 39.5 17.4 186
#> 3 Adelie Torgersen 40.3 18 195
#> 4 Adelie Torgersen NA NA NA
#> 5 Adelie Torgersen 36.7 19.3 193
#> 6 Adelie Torgersen 39.3 20.6 190
#> # ℹ 146 more rows
#> # ℹ 3 more variables: body_mass_g <int>, sex <fct>, year <int>| 항목 | 데이터 마스킹 | Tidy Evaluation |
|---|---|---|
| 정의 | - 데이터프레임 칼럼명을 직접적인 변수처럼 다룰 수 있는 능력. - $나 [[ ]] 없이 칼럼 참조를 단순화. |
- R 메타프로그래밍을 위한 프레임워크, 특히 tidyverse에서 사용. - 다양한 맥락에서 표현식을 캡처하고 평가하는 도구 제공. |
| 사용 사례 | - dplyr 함수에서 직접 데이터 조작. - 코드 가독성 향상. |
- 따옴표 없는 표현식으로 사용자 정의 함수 생성. - 표현식을 프로그래밍 방식으로 구성 및 평가. - 표현식 평가 맥락 제어. |
| 구현 | - 기본적으로 tidy evaluation 메커니즘을 사용하여 구현됨. | - rlang 패키지의 enquo(), quo(), !! 등을 사용. - 표현식과 그 환경을 캡처하기 위해 쿼저(Quosure)에 의존. |
| 복잡성 | - 최종 사용자를 위해 간소화. - 기본적인 복잡성을 추상화. |
- R 메타프로그래밍 이해 필요. - 고급 사용자에게 더 많은 유연성 제공. |
17.4 깔끔한 평가
깔끔한 평가(Tidy evaluation)은 R tidyverse 프레임워크로, 특히 비표준 평가(Non-Standard Evaluation, NSE)와 관련하여 tidyverse 함수로 프로그래밍하는 방법을 표준화했다. NSE는 R 함수가 표준과는 다른 맥락에서 표현식을 평가할 때 발생한다.
- 준인용(Quasiquotation):
enquo()함수를 사용하여 표현식을 캡처하고!!를 사용하여 표현식의 인용 제거(Unquoting)를 가능케 한다. - Pronouns (대명사):
.data대명사는 데이터프레임의 칼럼명을 명시적으로 참조하는 데 사용되어 모호성을 제거한다. - 함수:
enquo()는 표현식을 캡처하고,quo_name()은 표현식을 문자열로 변환하며,!!는 표현식 인용 제거 또는 주입 작업을 수행한다.
예를 들어, dplyr 패키지의 filter 및 select와 같은 동사를 사용하지만 함수에 칼럼명을 작성하려는 경우, 인수로 전달될 때 이러한 동사가 어떤 칼럼을 참조하는지 명확히 하기 위해 깔끔한 평가(tidy evaluation)가 사용된다.
library(dplyr)
library(palmerpenguins)
filter_and_select <- function(data, col_name, threshold) {
# 칼럼명 문자열을 기호로 변환
col_sym <- sym(col_name)
# 준인용(quasiquotation)을 사용해서 칼럼 표현식을 캡처
col_expr <- enquo(col_sym)
# !! 연산자를 이용하여 인용 제거(unquote)하고 표현식을 주입
data %>%
filter(!!col_expr > threshold) %>%
select(!!col_expr)
}
filter_and_select(penguins, "bill_length_mm", 55)
#> # A tibble: 5 × 1
#> bill_length_mm
#> <dbl>
#> 1 59.6
#> 2 55.9
#> 3 55.1
#> 4 58
#> 5 55.8sym() → enquo() → !!(뱅뱅) 구현 방식이 Defuse-and-Inject 패턴으로 내부 동작 방식은 동일하지만 사용자 구문은 { 칼럼명 }으로 깔끔해졌다.
- 데이터 마스킹: 변수를 사용하여 계산하는
arrange(),filter(),summarize()등의 함수에 사용. - Tidy-selection: 변수를 선택하는
select(),relocate(),rename()과 같은 함수에 사용.
17.4.1 그룹별 평균
데이터프레임의 요약통계량을 계산하는 코드를 작성해보자. 펭귄 종(species) 별로 부리 길이를 계산하는 코드를 작성해보자.
이번에는 그룹 변수와 데이터프레임 칼럼명을 달리하여 그룹별로 평균을 계산하는 함수를 작성하여 코드로 작성해보자.
get_group_mean <- function(dataframe, group_varname, varname) {
dataframe |>
group_by(group_varname) |>
summarise(부리길이_평균 = mean(varname, na.rm = TRUE))
}
get_group_mean(penguins, species, bill_length_mm)
#> Error in `group_by()`:
#> ! Must group by variables found in `.data`.
#> ✖ Column `group_varname` is not found.상기 코드가 동작하지 않는 이유는 함수에 tidyverse 코드를 단순히 전달해서 넣었기 때문에 발생했다. 다음과 같이 칼럼명을 포용(embracing)하는 방식으로 { 칼럼명 }과 같이 함수에 사용되는 데이터프레임 변수명을 명시적으로 작성할 경우 문제가 해결된다.
get_group_mean <- function(dataframe, group_varname, varname) {
dataframe |>
group_by( {{ group_varname }} ) |>
summarise(부리길이_평균 = mean( {{ varname }}, na.rm = TRUE))
}
get_group_mean(penguins, species, bill_length_mm)
#> # A tibble: 3 × 2
#> species 부리길이_평균
#> <fct> <dbl>
#> 1 Adelie 38.8
#> 2 Chinstrap 48.8
#> 3 Gentoo 47.517.4.2 그래프
데이터프레임에서 범주형 변수를 하나 선택하여 빈도수를 시각화하는 스크립트를 다음과 같이 작성한다.
범주형 변수를 막대그래프로 시각화하는 함수를 제작해보자.
함수 내에서 새로운 변수 이름을 생성하는 경우 := 연산자를 사용해야 한다. 깔끔한 평가(tidy evaluation)에서 =와 동일한 역할을 수행하는 것이 := 이기 때문이다. 예를 들어, 펭귄이 서식하고 있는 섬을 기준으로 빈도수를 내림차순 막대그래프를 작성할 경우, 함수 내부에서 범주형 변수를 다시 재정의해야 하는데 이때 := 연산자의 도입이 필요하다.
그래프를 작성할 때 거의 항상 등장하는 문제가 그래프 x-축, y-축 라벨을 붙이고 그래프 제목, 범례 등 텍스트를 넣어야 한다는 것이다. 이런 경우 stringr, glue 패키지의 다양한 함수를 깔끔한 평가(tidy evaluation)에서 지원하는 기능이 rlang 패키지의 englue() 함수다.
draw_bar_chart <- function(dataframe, varname, penguin_species) {
title_label <- rlang::englue("남극 서식 펭귄 종 ({{ varname }}) {penguin_species} 빈도수")
dataframe |>
filter({{ varname }} == penguin_species) |>
count( island ) |>
ggplot(aes(x = island, y = n )) +
geom_col(width = 0.5) +
labs(title = title_label)
}
penguins |> draw_bar_chart(species, "Adelie")17.5 요약
일반 프로그래밍 언어 함수와 달리 tidyverse의 데이터 과학 함수는 데이터 조작, 시각화, 모형 개발에 특화되어 있으며, 함수를 작성함으로써 코드의 가독성을 높이고 반복 작업을 줄일 수 있다.
데이터 과학 함수의 발전 과정에서 중요한 개념으로 데이터 마스킹과 깔끔한 평가를 꼽을 수 있다. 데이터 마스킹은 데이터프레임 칼럼을 직접 참조할 수 있게 해주는 기능으로, dplyr 패키지에서 핵심적인 역할을 한다. 깔끔한 평가는 tidyverse 함수로 프로그래밍할 때 비표준 평가와 관련된 문제를 해결하기 위한 프레임워크다.
dplyr의 group_by()와 summarise() 함수는 데이터프레임을 다루는 데 있어 중요한 역할을 하는데, 데이터프레임의 칼럼을 직접 참조할 수 있게 함으로써 코드의 가독성을 높이고 더 직관적인 코딩 경험을 제공한다. 그래프 생성에서도 ggplot2는 깔끔한 평가와 함께 데이터 마스킹 개념을 사용하여, 복잡한 코딩 과정 없이도 데이터프레임의 칼럼을 직접 참조하여 시각적 그래프를 직관적으로 생성할 수 있다.