들어가며
파머 펭귄 성별 분류 머신러닝 모델은 인구 관리 및 모니터링을 더 효율적으로 할 수 있기 때문에 중요합니다. 개별 펭귄의 성별을 정확하게 식별함으로써 펭귄 서식지에서 성비와 산란 패턴을 더 잘 이해하고 추적 할 수 있습니다. 이러한 정보는 지구온난화로 인한 기후위기 시대 펭귄생존을 보장하는 데 필수적입니다. 또한 성별 분류 모델은 노동집약적인 성별 식별 업무를 대폭 줄여 시간과 자원을 절약을 효율적으로 사용할 수 있도록 기여합니다.
데이터
데이터 입수
펭귄 데이터를 입수할 수 있는 방법은 크게 3가지가 있습니다.
데이터 패키지
로컬 csv 파일
클라우드 저장소: Microsoft Azure Blob Storage, Google Cloud Storage, DigitalOcean Spaces, Backblaze B2, Wasabi Hot Cloud Storage, Alibaba Cloud OSS, IBM Cloud Object Storage, Oracle Cloud Infrastructure Object Storage, OpenStack Object Storage (Swift), Rackspace Cloud Files
데이터 패키지
코드
library (palmerpenguins)<- palmerpenguins:: penguins#> # A tibble: 344 × 8 #> species island bill_length_mm bill_depth_mm flipper_…¹ body_…² sex year #> <fct> <fct> <dbl> <dbl> <int> <int> <fct> <int> #> 1 Adelie Torgersen 39.1 18.7 181 3750 male 2007 #> 2 Adelie Torgersen 39.5 17.4 186 3800 fema… 2007 #> 3 Adelie Torgersen 40.3 18 195 3250 fema… 2007 #> 4 Adelie Torgersen NA NA NA NA <NA> 2007 #> 5 Adelie Torgersen 36.7 19.3 193 3450 fema… 2007 #> 6 Adelie Torgersen 39.3 20.6 190 3650 male 2007 #> 7 Adelie Torgersen 38.9 17.8 181 3625 fema… 2007 #> 8 Adelie Torgersen 39.2 19.6 195 4675 male 2007 #> 9 Adelie Torgersen 34.1 18.1 193 3475 <NA> 2007 #> 10 Adelie Torgersen 42 20.2 190 4250 <NA> 2007 #> # … with 334 more rows, and abbreviated variable names ¹flipper_length_mm, #> # ²body_mass_g
$ pip install palmerpenguins
코드
import pandas as pdfrom palmerpenguins import load_penguins = load_penguins()#> species island bill_length_mm ... body_mass_g sex year #> 0 Adelie Torgersen 39.1 ... 3750.0 male 2007 #> 1 Adelie Torgersen 39.5 ... 3800.0 female 2007 #> 2 Adelie Torgersen 40.3 ... 3250.0 female 2007 #> 3 Adelie Torgersen NaN ... NaN NaN 2007 #> 4 Adelie Torgersen 36.7 ... 3450.0 female 2007 #> #> [5 rows x 8 columns]
csv 데이터
코드
library (tidyverse)<- read_csv ('data/penguins.csv' )#> # A tibble: 344 × 9 #> rowid species island bill_length_mm bill_dep…¹ flipp…² body_…³ sex year #> <dbl> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr> <dbl> #> 1 1 Adelie Torgersen 39.1 18.7 181 3750 male 2007 #> 2 2 Adelie Torgersen 39.5 17.4 186 3800 fema… 2007 #> 3 3 Adelie Torgersen 40.3 18 195 3250 fema… 2007 #> 4 4 Adelie Torgersen NA NA NA NA <NA> 2007 #> 5 5 Adelie Torgersen 36.7 19.3 193 3450 fema… 2007 #> 6 6 Adelie Torgersen 39.3 20.6 190 3650 male 2007 #> 7 7 Adelie Torgersen 38.9 17.8 181 3625 fema… 2007 #> 8 8 Adelie Torgersen 39.2 19.6 195 4675 male 2007 #> 9 9 Adelie Torgersen 34.1 18.1 193 3475 <NA> 2007 #> 10 10 Adelie Torgersen 42 20.2 190 4250 <NA> 2007 #> # … with 334 more rows, and abbreviated variable names ¹bill_depth_mm, #> # ²flipper_length_mm, ³body_mass_g
코드
import pandas as pd= pd.read_csv('data/penguins.csv' )print (palmer_df.head())#> rowid species island ... body_mass_g sex year #> 0 1 Adelie Torgersen ... 3750.0 male 2007 #> 1 2 Adelie Torgersen ... 3800.0 female 2007 #> 2 3 Adelie Torgersen ... 3250.0 female 2007 #> 3 4 Adelie Torgersen ... NaN NaN 2007 #> 4 5 Adelie Torgersen ... 3450.0 female 2007 #> #> [5 rows x 9 columns]
클라우드 저장소
코드
library (pins)library (Microsoft365R)<- Microsoft365R:: get_personal_onedrive ()<- board_ms365 (od, "krvote_board" )# board %>% pin_write(penguins) %>% pin_read ("penguins" )#> # A tibble: 344 × 8 #> species island bill_length_mm bill_depth_mm flipper_…¹ body_…² sex year #> <fct> <fct> <dbl> <dbl> <int> <int> <fct> <int> #> 1 Adelie Torgersen 39.1 18.7 181 3750 male 2007 #> 2 Adelie Torgersen 39.5 17.4 186 3800 fema… 2007 #> 3 Adelie Torgersen 40.3 18 195 3250 fema… 2007 #> 4 Adelie Torgersen NA NA NA NA <NA> 2007 #> 5 Adelie Torgersen 36.7 19.3 193 3450 fema… 2007 #> 6 Adelie Torgersen 39.3 20.6 190 3650 male 2007 #> 7 Adelie Torgersen 38.9 17.8 181 3625 fema… 2007 #> 8 Adelie Torgersen 39.2 19.6 195 4675 male 2007 #> 9 Adelie Torgersen 34.1 18.1 193 3475 <NA> 2007 #> 10 Adelie Torgersen 42 20.2 190 4250 <NA> 2007 #> # … with 334 more rows, and abbreviated variable names ¹flipper_length_mm, #> # ²body_mass_g
데이터 전처리
펭귄 데이터는 교육용으로 제공되지만 다른 데이터셋과 달리 결측값(NA)도 포함되어 있어 본격적인 분석을 위해서 결측값 현황을 정확히 확인하고 결측값에 대한 적절한 전략을 세우고 이를 처리해야 한다.
결측값 현황
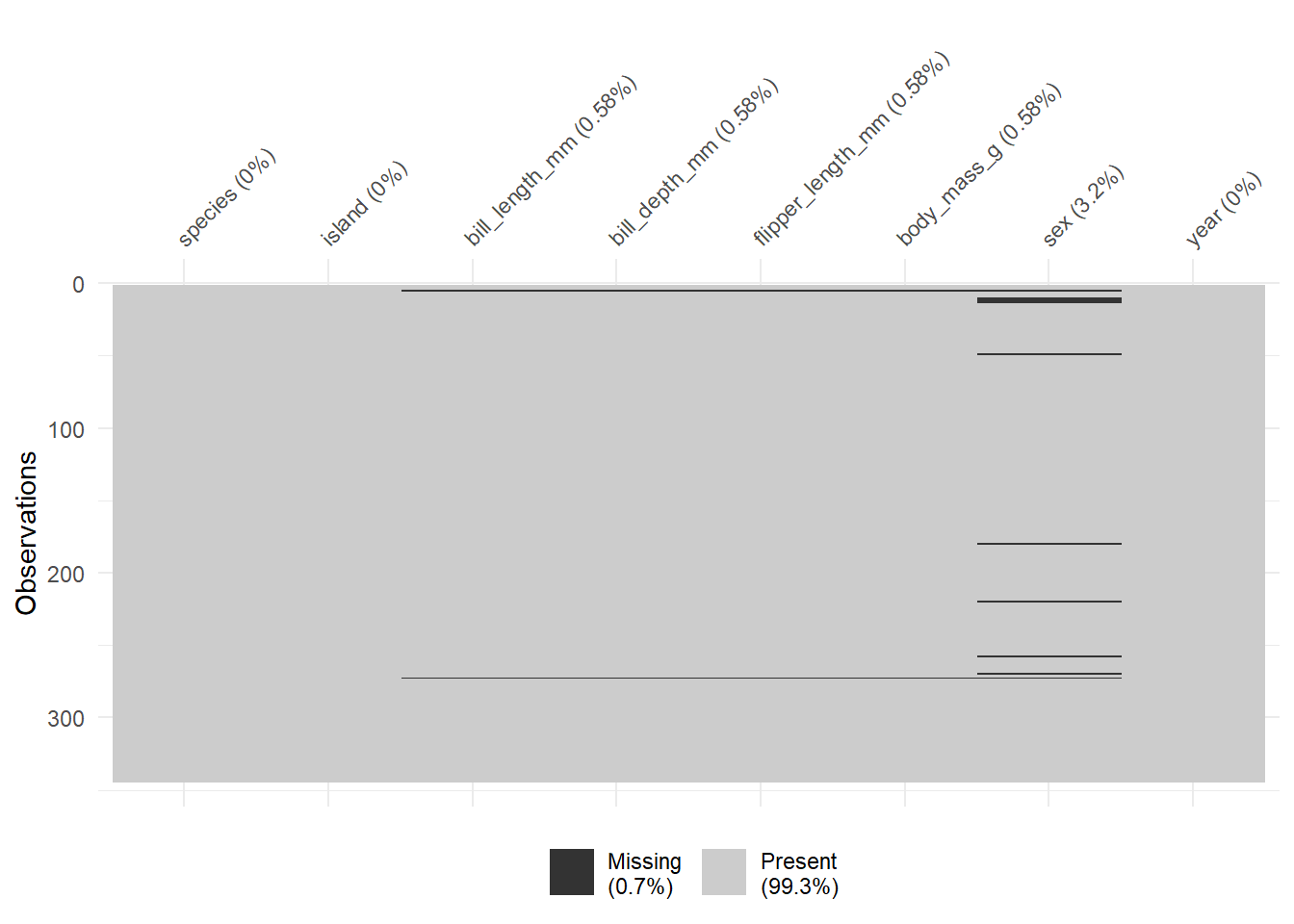
데이터프레임에 결측값이 어떻게 분포되어 있는지 먼저 시각적으로 파악하자. 상당수 결측값이 sex 칼럼에 몰려있는 것이 확인된다. 특히, 일부 펭귄의 경우 측정 변수가 모두 결측된 현황도 파악할 수 있다.
코드
library (tidyverse)library (naniar)library (palmerpenguins)<- palmerpenguins:: penguinsvis_miss (penguins_raw)
코드
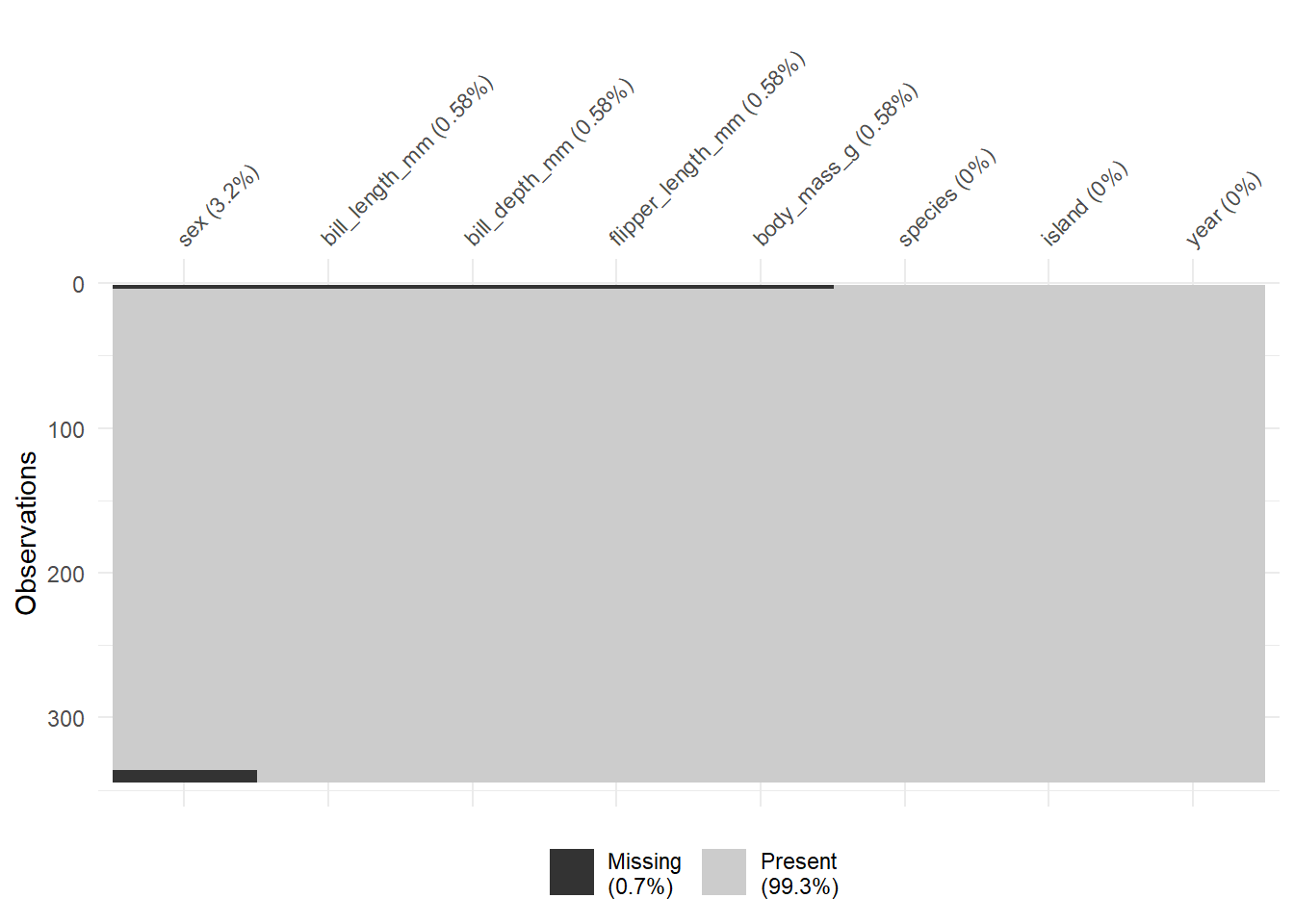
vis_miss (penguins_raw, cluster = TRUE , sort_miss = TRUE )
시각적으로 확인한 후에 기술통계량을 통해 정확한 결측값 현황을 확인해보자.
코드
nrow (penguins_raw) * ncol (penguins_raw)#> [1] 2752
코드
:: n_miss (penguins_raw)#> [1] 19
코드
:: n_complete (penguins_raw)#> [1] 2733
코드
miss_var_summary (penguins_raw)#> # A tibble: 8 × 3 #> variable n_miss pct_miss #> <chr> <int> <dbl> #> 1 sex 11 3.20 #> 2 bill_length_mm 2 0.581 #> 3 bill_depth_mm 2 0.581 #> 4 flipper_length_mm 2 0.581 #> 5 body_mass_g 2 0.581 #> 6 species 0 0 #> 7 island 0 0 #> 8 year 0 0
코드
miss_case_summary (penguins_raw)#> # A tibble: 344 × 3 #> case n_miss pct_miss #> <int> <int> <dbl> #> 1 4 5 62.5 #> 2 272 5 62.5 #> 3 9 1 12.5 #> 4 10 1 12.5 #> 5 11 1 12.5 #> 6 12 1 12.5 #> 7 48 1 12.5 #> 8 179 1 12.5 #> 9 219 1 12.5 #> 10 257 1 12.5 #> # … with 334 more rows
결측값 처리 전략
결측값 현황을 살펴보고 결측값이 많은 4번, 272번 펭귄은 분석에서 제거하고 sex 암수변수는 범주형 변수라 평균과 유사한 기능을 하는 최빈값(Mode) 정보를 이용하여 결측값을 채워넣는 것으로 한다.
코드
# get_mode <- function(x) { # ux <- unique(x) # ux[which.max(tabulate(match(x, ux)))] # } # # get_mode(penguins_csv$sex) <- penguins_raw %>% filter ( ! row_number () %in% c (4 , 272 ) ) %>% mutate ( sex = as.character (sex)) %>% mutate ( sex = if_else (is.na (sex), "male" , sex)) %>% mutate ( sex = factor ( sex, levels = c ("female" , "male" )))
탐색적 데이터 분석
결측값 제거 등을 통해 데이터 전처리 작업이 완료되었다. 다음 단계로 탐색적 데이터 분석을 통해 암수성별과 관련된 정보를 탐색적으로 파악해보자.
기술통계량
코드
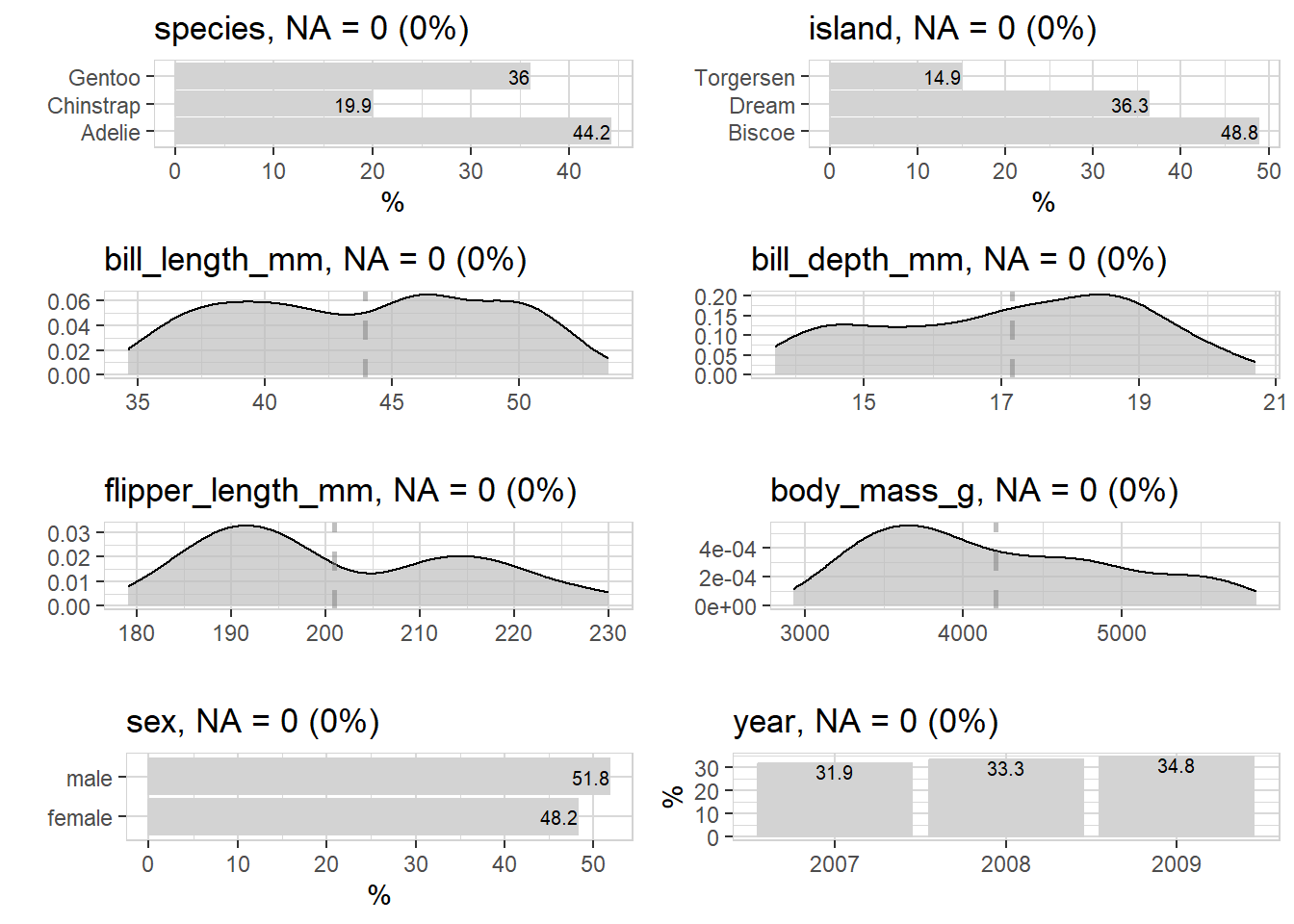
library (explore)%>% describe ()#> # A tibble: 8 × 8 #> variable type na na_pct unique min mean max #> <chr> <chr> <int> <dbl> <int> <dbl> <dbl> <dbl> #> 1 species fct 0 0 3 NA NA NA #> 2 island fct 0 0 3 NA NA NA #> 3 bill_length_mm dbl 0 0 164 32.1 43.9 59.6 #> 4 bill_depth_mm dbl 0 0 80 13.1 17.2 21.5 #> 5 flipper_length_mm int 0 0 55 172 201. 231 #> 6 body_mass_g int 0 0 94 2700 4202. 6300 #> 7 sex fct 0 0 2 NA NA NA #> 8 year int 0 0 3 2007 2008. 2009
단변량분석
코드
%>% explore_all ()
암수(target) 연관성
코드
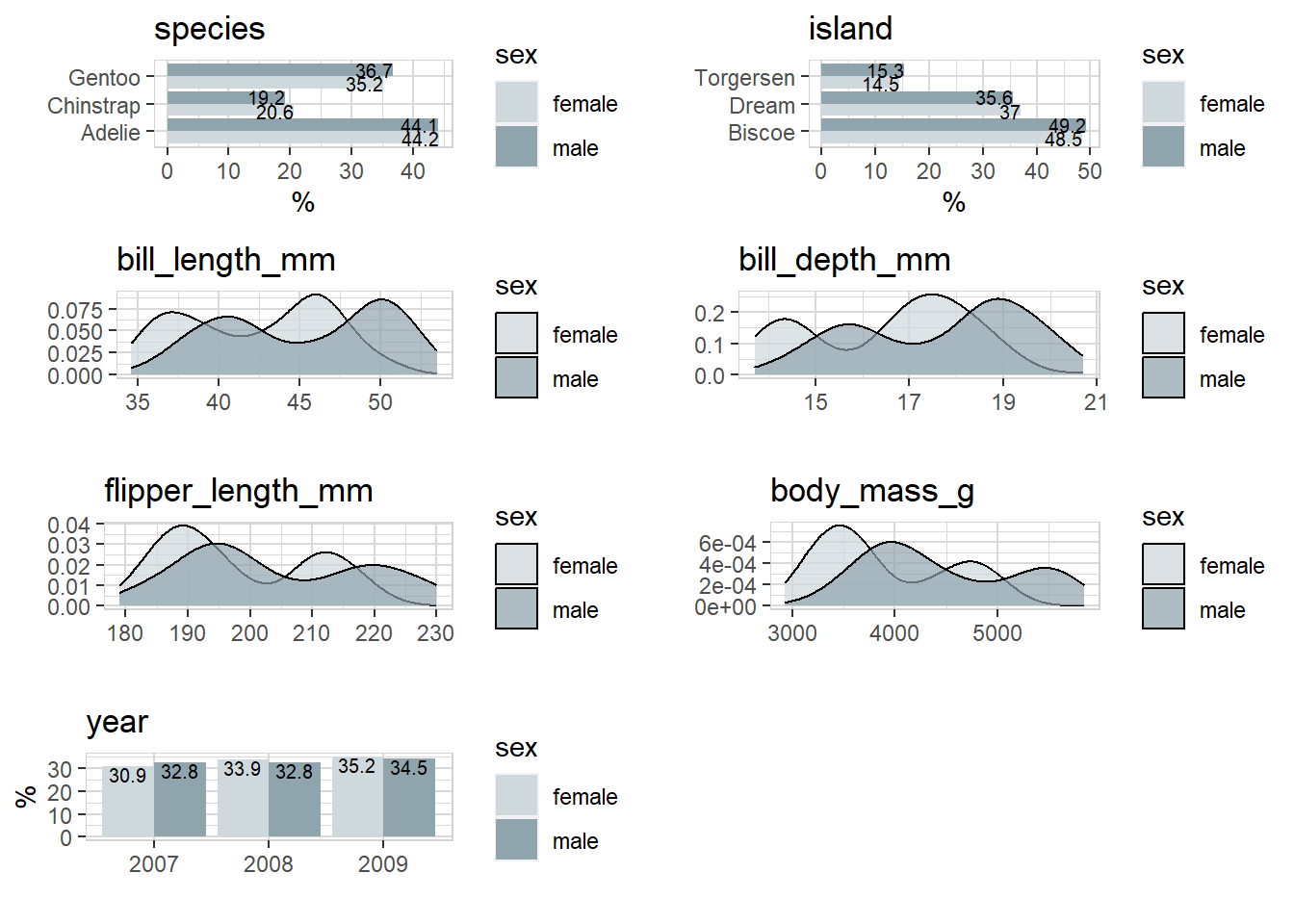
%>% explore_all (target = sex)
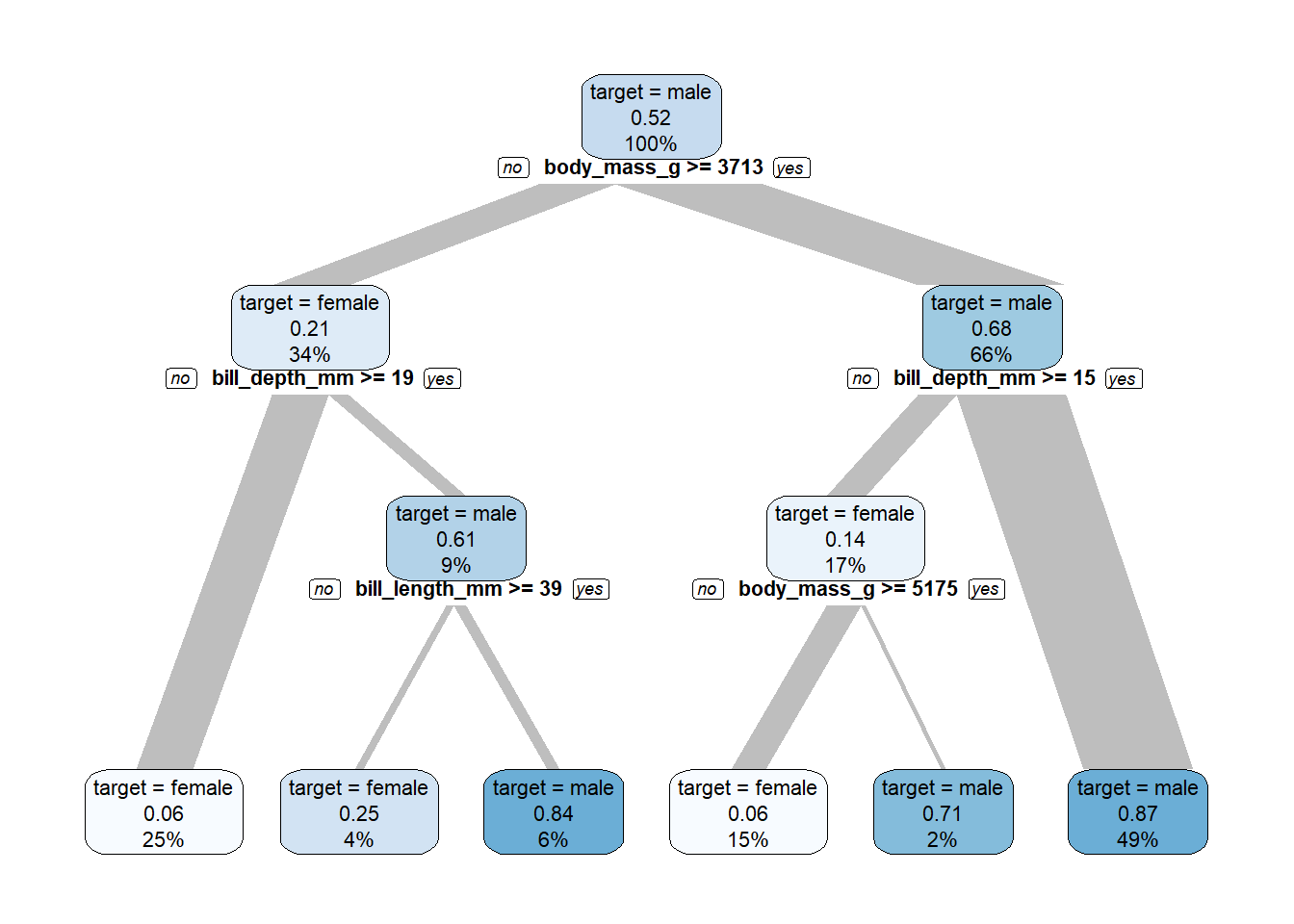
암수(target) 의사결정나무
코드
%>% explain_tree (target = sex)
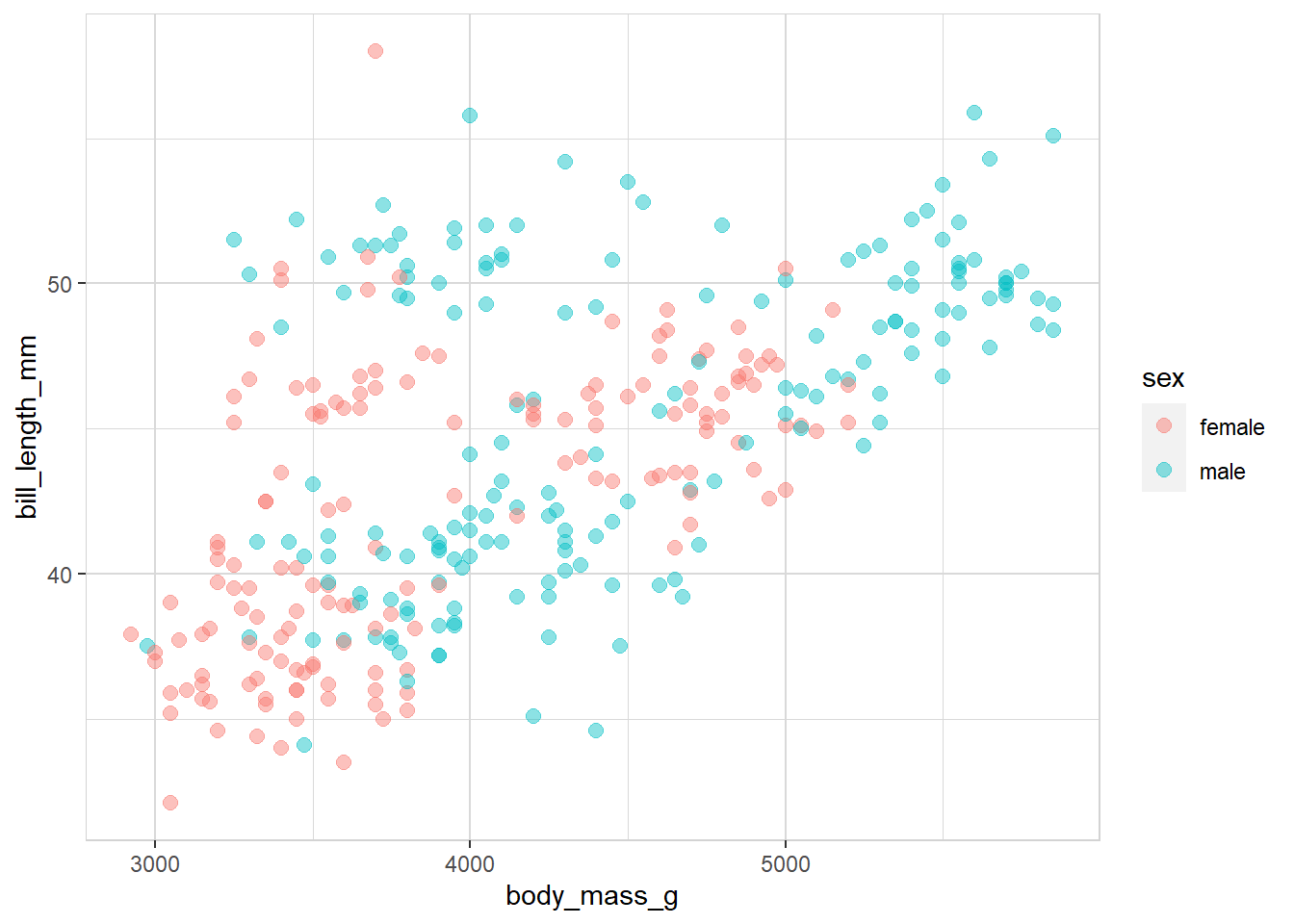
시각화
코드
%>% explore (body_mass_g, bill_length_mm, target = sex)
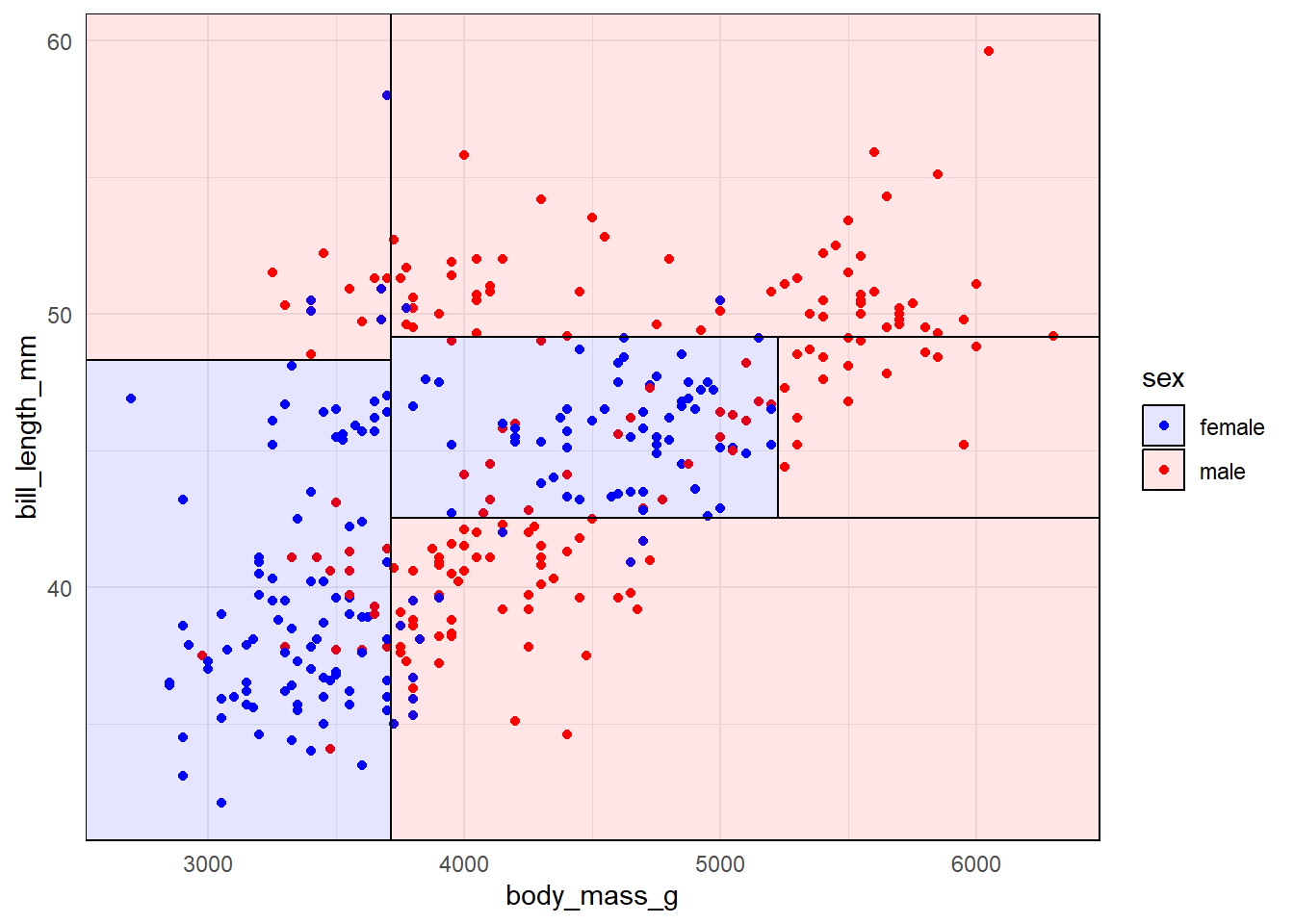
코드
library (tidymodels)library (parttree)<- decision_tree () %>% set_engine ("rpart" ) %>% set_mode ("classification" ) %>% fit (sex ~ body_mass_g + bill_length_mm, data = penguins_tbl)# 시각화 %>% ggplot (aes (x = body_mass_g, y = bill_length_mm)) + # geom_jitter(aes(col=species), alpha=0.7) + geom_point (aes (color = sex)) + geom_parttree (data = penguins_dt, aes (fill= sex), alpha = 0.1 ,flipaxes = FALSE ) + scale_color_manual (values = c ("female" = "blue" ,"male" = "red" )) + scale_fill_manual (values = c ("female" = "blue" ,"male" = "red" )) + theme_minimal ()
암수 분류 모형
훈련/시험 데이터셋
7:3으로 훈련시험 데이터셋을 나눈다.
코드
library (tidymodels)set.seed (123 )<- initial_split (penguins_tbl, strata = sex, prop = 0.7 )<- training (penguin_split)<- testing (penguin_split)set.seed (123 )<- bootstraps (penguin_train)#> # Bootstrap sampling #> # A tibble: 25 × 2 #> splits id #> <list> <chr> #> 1 <split [238/87]> Bootstrap01 #> 2 <split [238/85]> Bootstrap02 #> 3 <split [238/87]> Bootstrap03 #> 4 <split [238/86]> Bootstrap04 #> 5 <split [238/79]> Bootstrap05 #> 6 <split [238/83]> Bootstrap06 #> 7 <split [238/88]> Bootstrap07 #> 8 <split [238/83]> Bootstrap08 #> 9 <split [238/91]> Bootstrap09 #> 10 <split [238/87]> Bootstrap10 #> # … with 15 more rows
Feature Engineering
숫자형 변수에 대해서 정규화 작업을 진행하고 범주형 변수에 대해서 One-Hot 인코딩 작업을 수행하여 Feature 로 추가시킨다.
코드
<- recipe (sex ~ ., data = penguin_train) %>% step_corr (all_numeric (), threshold = 0.9 ) %>% step_normalize (all_numeric ()) %>% step_dummy (all_nominal (), - all_outcomes (), one_hot = TRUE )#> Recipe #> #> Inputs: #> #> role #variables #> outcome 1 #> predictor 7 #> #> Operations: #> #> Correlation filter on all_numeric() #> Centering and scaling for all_numeric() #> Dummy variables from all_nominal(), -all_outcomes()
기본모형(RF)
코드
<- rand_forest () %>% set_mode ("classification" ) %>% set_engine ("ranger" ) #> Random Forest Model Specification (classification) #> #> Computational engine: ranger
작업흐름
코드
<- workflow () %>% add_recipe (penguin_rec) %>% add_model (rf_spec)#> ══ Workflow ════════════════════════════════════════════════════════════════════ #> Preprocessor: Recipe #> Model: rand_forest() #> #> ── Preprocessor ──────────────────────────────────────────────────────────────── #> 3 Recipe Steps #> #> • step_corr() #> • step_normalize() #> • step_dummy() #> #> ── Model ─────────────────────────────────────────────────────────────────────── #> Random Forest Model Specification (classification) #> #> Computational engine: ranger
병렬처리 설정
코드
library (doParallel)<- parallel:: detectCores (logical = FALSE )<- makePSOCKcluster (cores)registerDoParallel (cores = cl)set.seed (77 )
훈련 적합
코드
:: tic ()<- penguin_wf %>% fit_resamples (resamples = penguin_boot,control = control_resamples (save_pred = TRUE )#> # Resampling results #> # Bootstrap sampling #> # A tibble: 25 × 5 #> splits id .metrics .notes .predictions #> <list> <chr> <list> <list> <list> #> 1 <split [238/87]> Bootstrap01 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 2 <split [238/85]> Bootstrap02 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 3 <split [238/87]> Bootstrap03 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 4 <split [238/86]> Bootstrap04 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 5 <split [238/79]> Bootstrap05 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 6 <split [238/83]> Bootstrap06 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 7 <split [238/88]> Bootstrap07 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 8 <split [238/83]> Bootstrap08 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 9 <split [238/91]> Bootstrap09 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> 10 <split [238/87]> Bootstrap10 <tibble [2 × 4]> <tibble [0 × 3]> <tibble> #> # … with 15 more rows :: toc ()#> 6.75 sec elapsed
모델 평가
코드
collect_metrics (rf_rs)#> # A tibble: 2 × 6 #> .metric .estimator mean n std_err .config #> <chr> <chr> <dbl> <int> <dbl> <chr> #> 1 accuracy binary 0.887 25 0.00746 Preprocessor1_Model1 #> 2 roc_auc binary 0.946 25 0.00396 Preprocessor1_Model1
오차 행렬
코드
%>% conf_mat_resampled () %>% pivot_wider (names_from = Truth, values_from = Freq)#> # A tibble: 2 × 3 #> Prediction female male #> <fct> <dbl> <dbl> #> 1 female 37.7 4.88 #> 2 male 4.84 38.4
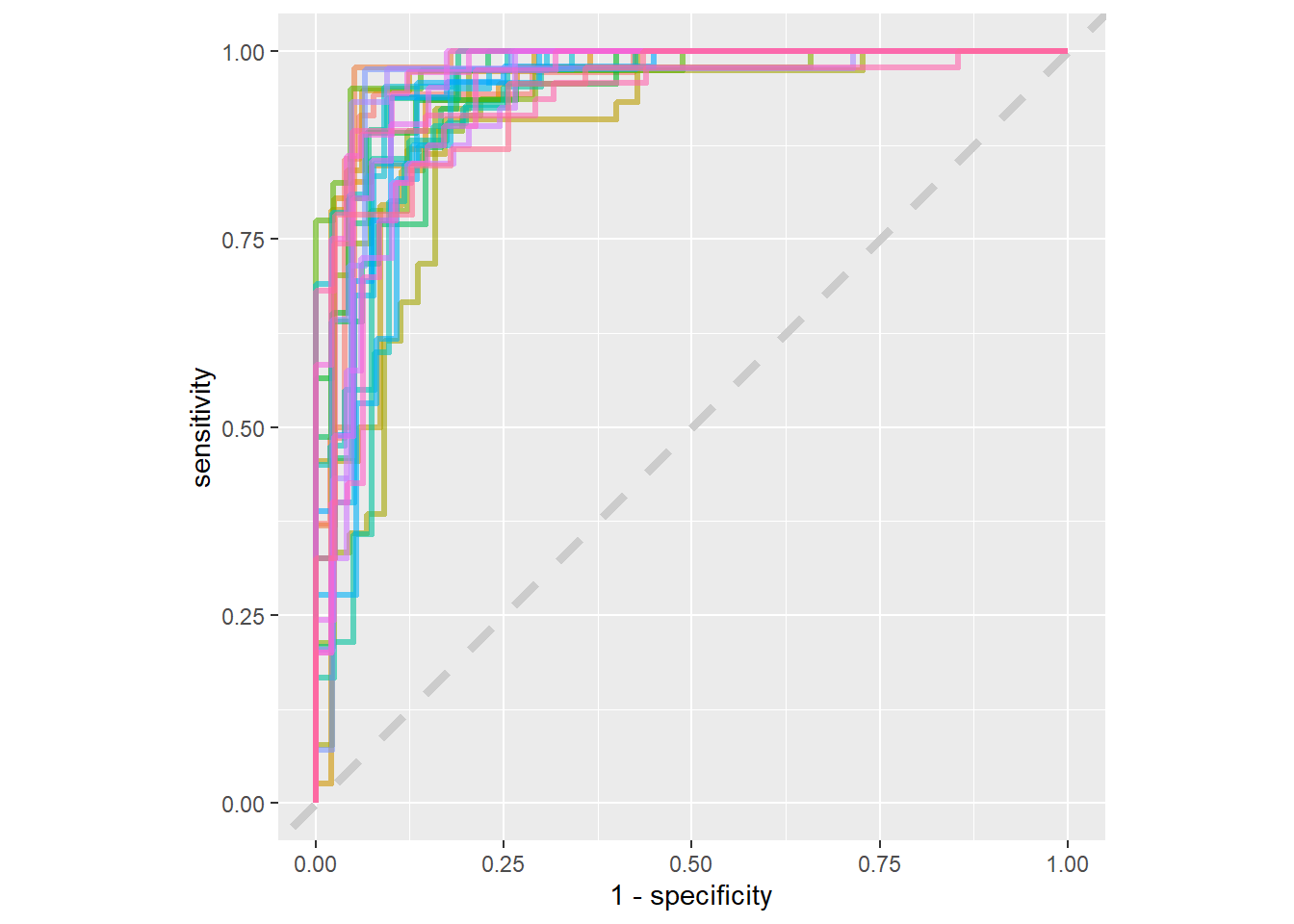
ROC 곡선
코드
%>% collect_predictions () %>% group_by (id) %>% roc_curve (sex, .pred_female) %>% ggplot (aes (1 - specificity, sensitivity, color = id)) + geom_abline (lty = 2 , color = "gray80" , size = 1.5 ) + geom_path (show.legend = FALSE , alpha = 0.6 , size = 1.2 ) + coord_equal ()
최종 모형
코드
<- penguin_wf %>% last_fit (penguin_split)#> # Resampling results #> # Manual resampling #> # A tibble: 1 × 6 #> splits id .metrics .notes .predictions .workflow #> <list> <chr> <list> <list> <list> <list> #> 1 <split [238/104]> train/test split <tibble> <tibble> <tibble> <workflow>
모형 성능
코드
<- collect_metrics (penguin_final)
암수 성별 예측모형의 최종 모형 정확도는 0.8557692 이고 ROC 는 0.9377778 이다.
코드
collect_predictions (penguin_final) %>% conf_mat (sex, .pred_class)#> Truth #> Prediction female male #> female 41 6 #> male 9 48
실제 암컷 펭귄을 수컷 펭귄으로 오분류하는 것이 그 반의 경우보다 30% 정도 더 높게 나온다.
설명가능한 ML
DALEX
코드
library (DALEX)library (DALEXtra) library (modelStudio) # install_dependencies() <- penguin_wf %>% fit (data = penguin_train)<- as.numeric (penguin_train$ sex)<- explain_tidymodels (penguin_fit,data = penguin_train,y = y_penguin,label = "Penguin RF" )set.seed (123 )<- penguin_train %>% slice_sample (n = 3 )modelStudio (penguin_explainer, new_observation = new_penguins)
주요 기여사항
파머 펭귄 성별 분류 머신러닝 모델은 인구 관리 및 모니터링을 더 효율적으로 할 수 있는 예측 분류 모형을 개발하였다. 예측 모형의 정확도는 0.8557692 으로 기존 통계모형보다 우수한 정확도를 보이고 있다.
한계
예측 모형의 정확도가 0.8557692 으로 기존 통계모형보다 높지만 실제 업무에 배포하여 적용하는 기준 정확도 95% 에는 미치지 못하는 한계가 있고, 개발된 암수분류 모형을 실제 운영환경에서 사용할 수 있는 추가 작업이 필요한 것으로 확인되었습니다.
결론
개별 펭귄의 성별을 정확하게 식별함으로써 펭귄 서식지에서 성비와 산란 패턴을 더 잘 이해하고 추적 할수 있는 성별 예측 기계학습 모형을 개발하였고 이를 실제 업무에 적용하기 위한 도전과제도 추가로 발굴되었다. 금번 프로젝트가 후속 연구개발로 이어져 기후 온난화로 인한 안정적인 펭귄 개체수 유지를 위한 발판으로 이어질 것으로 기대된다.